Wprowadzenie
Pierwsza lekcja była wprowadzeniem do korzystania z API Wellcome Collection z pomocą języka R. Udało nam się pozyskać jedną stronę wielostronicowej listy obiektów - zdigitalizowanych prac XVI-wiecznego rytownika Josta Ammana. Dane dostarczone przez serwer muzeum miały złożoną strukturę, jednak ostatecznie udało się nam wydobyć z niej wybrane metadane prac Ammana, przechowywanych w Wellcome. Niestety, były to dane tylko 10 ze 113 obiektów przypisywanych temu autorowi - API Wellcome zwróciło jedynie pierwszą stronę wyników wyszukiwania.
W niniejszej lekcji postaramy się pozyskać wszystkie dane o interesujących nas obiektach, przefiltrować je tak, aby zostały w nich wyłącznie informacje o obiektach będących drzeworytami i pobrać na dysk cyfrowe wizerunki tych obiektów.
Cele lekcji
Celem lekcji jest wdrożenie do pracy z API instytucji dziedzictwa z wykorzystaniem metod języka R oraz zdobycie umiejętności korzystania z platformy Posit.cloud, pozwalającej na pracę w R bezpośrednio w przeglądarce, bez konieczności instalowania czegokolwiek na własnym komputerze.
Efekty
- praktyka pracy z API instytucji dziedzictwa,
- podstawy budowania pętli i funkcji w R,
- podstawy pracy z ramkami danych w R,
- podstawy pobierania plików i zarządzania plikami w R.
Wymagania
Do korzystania z lekcji konieczne są:
- przećwiczona lekcja numer I,
- przeglądarka internetowa,
- założenie darmowego konta na platformie Posit.cloud.
Posit.cloud to produkt firmy odpowiedzialnej za RStudio - powszechnie wykorzystywany edytor i środowisko programistyczne dla języka R.
Część merytoryczna
Podzielmy naszą pracę na trzy etapy. W pierwszym nauczymy się, jak automatycznie pobrać wszystkie dane dotyczące interesujących nas artefaktów. W drugim - odpowiednio przefiltrujemy pozyskane dane, a w trzecim - automatycznie pobierzemy cyfrowe wizerunki interesujących nas prac.
Zastosujmy pętlę
Jak pamiętamy, udało nam się pobrać jedynie pierwszą stronę wyników wyszukiwania. W strukturze tych danych znajduje się klucz nextPage, który podaje adres kolejnej strony wyników. Jeśli wyświetlimy ten nowy adres w przeglądarce, serwer znów poda nam kolejną porcję danych i adres kolejnej strony wyników. Szybkie porównanie tych adresów pozwala zobaczyć, że różnią się one parametrem page=:
https://api.wellcomecollection.org/catalogue/v2/works?query=Amman+Jost&page=1
https://api.wellcomecollection.org/catalogue/v2/works?query=Amman+Jost&page=2
https://api.wellcomecollection.org/catalogue/v2/works?query=Amman+Jost&page=3
https://api.wellcomecollection.org/catalogue/v2/works?query=Amman+Jost&page=4
https://api.wellcomecollection.org/catalogue/v2/works?query=Amman+Jost&page=5
…
https://api.wellcomecollection.org/catalogue/v2/works?query=Amman+Jost&page=12Chcemy zebrać wszystkie dane o pracach Josta - czy to oznacza, że musimy aż dwanaście razy wykonać wszystkie czynności opisane w lekcji I? Oczywiście, że nie! Takie podejście byłoby może racjonalne przy danych o niewielkiej objętości, ale zupełnie nieefektywne przy danych publikowanych na dziesiątkach czy setkach stron.
Zamiast robić powtarzalne rzeczy, lepiej oddać to oprogramowaniu - skorzystajmy z pętli (loop). Pętla w programowaniu to taki fragment kodu, który powtarza określone czynności zanim nie zostanie spełniony określony warunek.
Gdybyśmy chcieli ręcznie pracować z dwunastoma stronami wyników naszego wyszukiwania w bazach danych Wellcome Collection, musielibyśmy wykonać dwanaście razy tę samą czynność:
- połączyć się z API i pobrać dane
- dodać wiersze do wyjściowej ramki danych
To jednak trochę powtarzalnej pracy - niech program zrobi to za nas! Musimy tylko zdecydować, jaki podać mu warunek, tak aby pętla nie wykonywała się w nieskończoność. Kiedy wysłaliśmy do API Wellcome Collection pierwszy request, serwer zwrócił odpowiedź zawierającą nie tylko metadane prac Ammana, ale też dodatkowe informacje. W kluczu totalResults znalazla się informacja, że mamy dostęp do 113 zdigitalizowanych prac:
{
"type":"ResultList",
"pageSize":10,
"totalPages":12,
"totalResults":113,
"results":[...]
}Przy każdym żądaniu nasza ramka danych zwiększy się o kolejne obiekty - musi być ich ostatecznie 113. Wykorzystajmy ten warunek w budowie pętli.
Budujemy pętlę
Zobaczmy taki kod w języku R:
x <- 0
while(x != 10) {
x <- x + 1
print(x)
}Pętla while wykonuje określony blok kodu, dopóki warunek logiczny jest spełniony . Po każdej iteracji (każdorazowym wykonaniu bloku kodu) warunek jest sprawdzany. Jeśli nadal jest spełniony, pętla kontynuuje działanie - jeśli nie, pętla się zatrzymuje.
W kodzie podanym wyżej warunkiem, którego prawdziwość sprawdzana jest za każdą iteracją, jest x różne od 10. Początkowo wartość x to 0, a każda kolejna iteracja zwiększa tę wartość o 1. Funkcja print() wyświetla wartość x. Po dziesięciu wykonaniach tego samego kodu pętla się zatrzymuje - x ma ostatecznie wartość 10, więc warunek x != 10 nie jest już spełniony.
Ten schemat możemy zastosować w naszym kodzie. Zdefiniujemy obiekt total, który będzie zawierał informację o liczbie wierszy w ramce danych. Za każdą iteracją będziemy dodawać kolejne wiersze do ramki danych (najpierw będzie ich 0, potem 10, potem 20 itd.). Warunkiem sprawdzanym przy każdej iteracji będzie total != 113, bo właśnie na tyle obiektów czekamy.
Nie jest to oczywiście specjalnie skomplikowane rozwiązanie, ma też swoje wady. Jeśli w przypadku jakiegoś błędu w komunikacji między naszym programem a API jakaś część danych się nie dogra, pętla może wykonywać się w nieskończoność (warunek total != 113 będzie wciąż spełniany). Dlatego zmodyfikujmy warunek na total < 113 - unikniemy tym samym tego niebezpieczeństwa. Jeśli liczba pozyskanych obiektów przestanie być mniejsza niż liczba wszystkich obiektów dostępnych w API, pętla się zatrzyma.
Elementem naszego programu musi być też obiekt page, w którym trzymać będziemy numer strony. Niech zwiększa się o 1 z każdą iteracją pętli. Brakuje nam jeszcze jednego obiektu - docelowej ramki danych, w której znajdą się informacje o wszystkich pozyskanych dziełach Ammana Josta.
Czytając przykładowy kod naszej pętli warto wrócić do pierwszej lekcji na temat Wellcome Collection i przypomnieć sobie metodę wysyłania i odczytywania requestów do API. Korzystamy z pakietu jsonlite i dostępnej w nim metody fromJSON().
W kodzie naszej pętli pojawią się nowe elementy. Funkcja paste0() łączy dwa ciągi tekstowe (bez spacji pomiędzy nimi) - zależy nam na tym, aby dynamicznie budować adresy poszczególnych stron żądania:
page < -1
api_url <- 'https://api.wellcomecollection.org/catalogue/v2/works?query=Amman%20Jost&page='
current_api_url <- paste0(api_url,page)Pojawi się też funkcja rbind(), która pozwala na łączenie ze sobą ramek danych. Każdą listę obiektów zwracaną przez API przetwarzamy jako ramkę danych dzięki funkcji fromJSON() z atrybutem flatten = TRUE, spłaszczającym strukturę oryginalnego JSONa do postaci dwuwymiarowej tabeli.
Funkcja rbind() jest bardzo przydatna, ale wymaga od nas, żeby ramki, które będziemy ze sobą łączyć, miały tę samą ilość kolumn. Okazuje się, że w Wellcome Collection to nie takie pewne. Przykładowo, do piątej strony wyników wyszukiwania w API dostępna jest kolumna lettering, której brakuje już w schemacie opisu obiektów na szóstej stronie wyników. W takim przypadku funkcja rbind() zwróci nam błąd - ramki danych, które łączymy, powinny mieć taką samą strukturę.
Żeby tego uniknąć, skorzystajmy z wykorzystywanej już w poprzedniej lekcji funkcji z pakietu dplyr i od razu wybierzmy tylko interesujące nas kolumny.
Kod źródłowy pętli
# 1. Wczytujemy dodatkowe pakiety R, aby skorzystać z metod przez nie oferowanych.
library(jsonlite)
library(dplyr)
# 2. Przypisujemy do api_url podstawowy adres, na który będziemy wysyłać żądanie. W adresie brakuje informacji o stronie - tę dodawać będziemy dynamicznie.
api_url <- 'https://api.wellcomecollection.org/catalogue/v2/works?query=Amman%20Jost&page='
# 3. Tworzymy obiekty, które będą nadpisywane przez pętlę.
# total - liczba wyników (na początku 0, docelowo 113)
# page - liczba stron - na początku 1
# objects - pusta ramka danych, w której znajdą się metadane wyszukiwanych obiektów
total <- 0
page <- 1
objects <- data.frame()
# 4. Konstruujemy pętlę z warunkiem, którego elementem jest dynamicznie zmieniana wartość total
while(total < 113) {
# 4.1 Budujemy dynamicznie adres żądania i go wyświetlamy
current_api_url <- paste0(api_url,page)
cat(paste0('\n', current_api_url))
# 4.2 wysyłamy żądanie i spłaszczamy strukturę danych
request <- fromJSON(paste0(current_api_url), flatten = TRUE)
# 4.3 Wybieramy tylko niektóre kolumny z ramki danych i tworzymy z nich ramkę current_objects
current_objects <- request$results %>% select(id, workType.label, title, description, thumbnail.url, thumbnail.license.label)
# 4.4 Dopisujemy ramkę current_objects do naszej wyjściowej ramki (objects)
objects <- rbind(objects, current_objects)
# 4.5 modyfikujemy wartość wektora total, tak aby pętla mogła się kiedyś skończyć :)
total <- nrow(objects)
# 4.6 aby wysłać żądanie o kolejną stronę wyników, zwiększamy numer strony
page <- page + 1
}W kodzie pojawiła się też funkcja nrow() zwracająca liczbę wierszy ramki danych - to kluczowy element, bo wykorzystywany w warunku naszej pętli. Funkcja cat() wyświetla w konsoli informację o adresie aktualnego żądania - ciąg tekstowy \n oznacza, że wyświetlać się będzie od nowej linii.
Po uruchomieniu pętli otrzymamy 113 wyników wyszukiwania. Ale być może ta liczba zmieni się do czasu, w którym zajrzycie na tę lekcję. Zbiory muzeum mogą przyrastać. Spróbujmy przebudować naszą pętlę tak, aby była bardziej uniwersalna.
Ładniej i wygodniej - zbudujmy własną funkcję
Funkcja w programowaniu to taka struktura kodu, która ma wykonać określone zadanie lub operację na danych wejściowych, przekazywanych do funkcji jako argumenty. Dzięki funkcjom możemy uniknąć pisania wciąż tego samego kodu i łatwiej zarządzać własnym programem.
Bardzo prosta funkcja w R wygląda tak:
add10 <- function(i) {
return(i + 10)
}i działa tak:
> add10(100)
[1] 110Nietrudno zgadnąć, że celem funkcji add10() jest dodanie 10 do liczby podanej jako argument. Warto zwrócić uwagę na słowo function() oraz i, które jest tutaj argumentem - nie definiujemy go, bo użytkownik czy użytkowniczka funkcji może sama to zrobić.
Spróbujmy przepisać naszą pętlę do funkcji, która ułatwi nam wysyłanie żądań do Wellcome Collection API:
getDataFromWC <- function(s = "Amman Jost", cols = c("id", "workType.label", "title", "description", "thumbnail.url", "thumbnail.license.label")) {
# 1. Funkcja przyjmuje dwa atrybuty: frazę wyszukiwania (s) oraz listę kolumn (metadanych)
# 2. Potrzebujemy dwóch bibliotek: jsonlite do łączenia się z API, a dplyr do opracowania pozyskanych danych
library(jsonlite)
library(dplyr)
# 3. Formatujemy frazę wyszukiwania (m.in. pozbywamy się spacji na rzecz %20)
s <- URLencode(s)
# 4. Dynamicznie budujemy adres żądania, łącząc endpoint z frazą wyszukiwania
api_url <- paste0('https://api.wellcomecollection.org/catalogue/v2/works?query=',s,'&page=')
# 5. Definiujemy podstawowe zmienne (wektory), które będą się nadpisywać w trakcie działania pętli
# total - liczba pozyskanych obiektów (wierszy)
# page - numer strony
total <- 0
page <- 1
# 6. Musimy sprawdzić liczbę wszystkich dostępnych obiektów
# Zapisujemy ją do total_results
first <- fromJSON(paste0(api_url, 1), flatten = TRUE)
total_results <- first$totalResults
# 7. Wyświetlamy informację o zakładanej liczbie obiektów
cat(paste0('\n Total results: ', total_results))
# 8. Tworzymy pustą ramkę danych, do której dogrywać będziemy wiersze z informacjami o kolejnych obiektach
objects <- data.frame()
# 9. Definiujemy pętlę, która będzie wykonywać się do czasu, aż liczba pozyskanych obiektów (total) będzie równa liczbie dostępnych obiektów w API (total_results)
while(total < total_results) {
# 9.1 dynamicznie definiujemy aktualny adres żądania (zmienia się strona)
current_api_url <- paste0(api_url,page)
# 9.2 Wyświetlamy informację o tym adresie i numerze strony
cat(paste0('\n', current_api_url))
cat(paste0('\n', page))
# 9.3 Wysyłamy żądanie do API
request <- fromJSON(paste0(current_api_url), flatten = TRUE)
# 9.4 Z ramki danych dostępnej w odpowiedzi serwera pobieramy wybrane kolumny (metadanych)
current_objects <- request$results %>% select(all_of(cols))
# 9.5 Dodajemy nowe wiersze do zapisanych już danych
objects <- rbind(objects, current_objects)
# 9.6 Sprawdzamy liczbę pozyskanych obiektów
total <- nrow(objects)
# 9.7 Zwiększamy numer strony o 1
page <- page + 1
# 9.8 Czekamy 3 sekundy
Sys.sleep(3)
}
# 10. Zwracamy pozyskane dane w postaci ramki danych objects
return(objects)
}Funkcja getDataFromWC() przyjmuje dwa argumenty. Pierwszy to fraza wyszukiwania, drugi - lista kolumn (metadanych), które mają być zapisane. Listę kolumn podajemy za pomocą funkcji c() tworzącej wieloelementowy wektor. To, że konkretną wartość argumentów podajemy już w kodzie funkcji oznacza, że będą traktowane jako domyślne. Konstrukcja naszej funkcji jest o tyle uniwersalna, że te domyślne argumenty możemy łatwo nadpisać i wyszukiwać obiekty innych autorów niż Amman Jost i pozyskiwać z tych obiektów inne metadane.
W kodzie naszej funkcji pojawia się URLencode() - to funkcja, która przekształca ciąg tekstowy do postaci przyjaznej dla adresów URL - w końcu praca z API jest pracą z adresami URL. Adresy te jak wiadomo nie mogą mieć spacji - dlatego ciąg tekstowy Amman Jost zamieni się na Amman%20Jost.
Kolejna nowość to all_of() wykorzystane w funkcji select(). Funkcja select() oczekuje, że przekazane jej argumenty będą nazwami kolumn - bez użycia all_of() select potraktowałaby wysłaną tam zmienną jako pojedynczą nazwę kolumny.
Do naszej funkcji włożyliśmy też Sys.sleep(3) - tym razem to funkcja, która na trzy sekundy zatrzyma wykonywanie programu, a więc kolejną iterację i wysłanie kolejnego żądania. Nie chcemy, żeby serwer API zablokował nas za zbyt intensywne pozyskiwanie kolejnych danych.
Funkcję kończy return(objects) - pozyskane dane udostępniamy na zewnątrz funkcji, dzięki czemu możemy je przypisać do jakiegoś obiektu R.
Przygotowana przez nas funkcja ma niestety jedną wadę. Wyobraźmy sobie, że pobieramy bardzo dużą liczbę obiektów i funkcja przechodzi przez dziesiątki czy setki stron wyników wyszukiwania. Wszystko działa bardzo dobrze, ale nagle - przez jakąś awarię - tracimy dostęp do internetu. Także serwer API może być tymczasowo niedostępny, albo w zwracanych przez niego wynikach nie ma oczekiwanej przez nas kolumny. W takim przypadku praca funkcji zostaje przerwana i zostajemy z niczym - żadne dane się nie zapisują. To, jak zabezpieczyć się przed takim efektem niespodziewanych błędów, omówimy już przy okazji innej lekcji.
Aby skorzystać z funkcji, wywołujemy ją przy deklaracji nowego obiektu:
our_data <- getDataFromWC()Filtrujemy dane i pobieramy pliki
Ostatnią częścią naszej lekcji jest przefiltrowanie pozyskanych danych tak, aby zostały w nich wyłącznie drzeworyty oraz pobranie ich cyfrowych reprodukcji.
Nasza ramka danych zawiera sześć kolumn - wyświetlimy je funkcją names():
> names(our_data)
[1] "id" "workType.label" "title" "description"
[5] "thumbnail.url" "thumbnail.license.label"Interesować nas będzie teraz kolumna workType.label, zawierająca informacje o typie obiektu. Bardzo łatwo sprawdzić, jakie wartości są w niej dostępne - korzystamy z funkcji table(), która policzy wystąpienia poszczególnych wartości w kolumnie:
> table(our_data$workType.label)
Archives and manuscripts Books Digital Images Pictures
1 17 1 94 Przefiltrujmy teraz nasze dane tak, aby zostały w nich jedynie obiekty o typie pictures. Korzystamy z pakietu dplyr, który musimy włączyć, aby mieć możliwość skorzystania z jego metod:
library(dplyr)
filtered_data <- our_data %>% filter(workType.label == 'Pictures')Rzut oka na ramkę danych wystarczy aby zauważyć, że nie wszystkie obiekty mają adres URL w kolumnie thumbnail.url. Musimy filtrować jeszcze raz, tym razem za pomocą funkcji is.na(), która sprawdza, czy dana wartość jest czy nie jest ustalona. NA (Not Available) reprezentuje brak danych lub wartość nieokreśloną. Jest to specjalna wartość używana do oznaczania braku danych w ramkach danych, wektorach lub innych strukturach danych.
filtered_data <- filtered_data %>% filter(!is.na(thumbnail.url))Znak wykrzyknika to zaprzeczenie: interesują nas wyłącznie te obiekty, których wartość metadanych thumbnail.url jest ustalona. Warto zauważyć, że nadpisaliśmy też naszą przefiltrowaną ramkę danych (filtered_data).
Pozostaje nam tylko pobrać grafiki, odwołując się do adresów z kolumny filtered_data. Możemy zrobić to za pomocą funkcji, która dodatkowo nada im odpowiednie nazwy.
getFilesFromWC <- function(df) {
# 1. liczymy iteracje
iteration <- 1
# 2. pętla for - przechodzimy przez wszystkie wiersze (wartości) w kolumnie thumbnail.url naszej ramki danych
for(image_url in df$thumbnail.url) {
# 3. tworzymy nazwę pliku z kolumny id, przy czym musimy wskazać numer wiersza
fname <- paste0(df$id[iteration],'.jpg')
# 3.1 wyświetlamy sobie adres i docelową nazwę pliku
cat(paste0('\n', image_url, ' -- ', fname))
# 3.2 pobieramy plik, plik docelowy (destfile) to dynamicznie generowana nazwa pliku
download.file(url = image_url, destfile = fname)
# 3.3 doliczamy kolejną iterację
iteration <- iteration + 1
# 3.4 trzy sekundy przerwy
Sys.sleep(3)
}
} Jako argument funkcji przekażemy naszą przefiltrowaną ramkę danych. W poprzedniej lekcji korzystaliśmy już ze znaku dolara ($) pozwalającego na wybór kolumny w ramce danych. Teraz dodajemy do tego jeszcze indeks (czy też numer wiersza) według schematu:
ramka.danych$kolumna[numer_wiersza]Zastosowaliśmy też znowu pętlę - jednak tym razem for a nie while. Pętla for służy do iteracji po zbiorze elementów - jeśli w naszej przefiltrowanej ramce danych mieliśmy 86 wierszy, pętla for przejdzie przez nie wszystkie i zatrzyma się po wykonaniu określonych czynności na ostatnim wierszu.
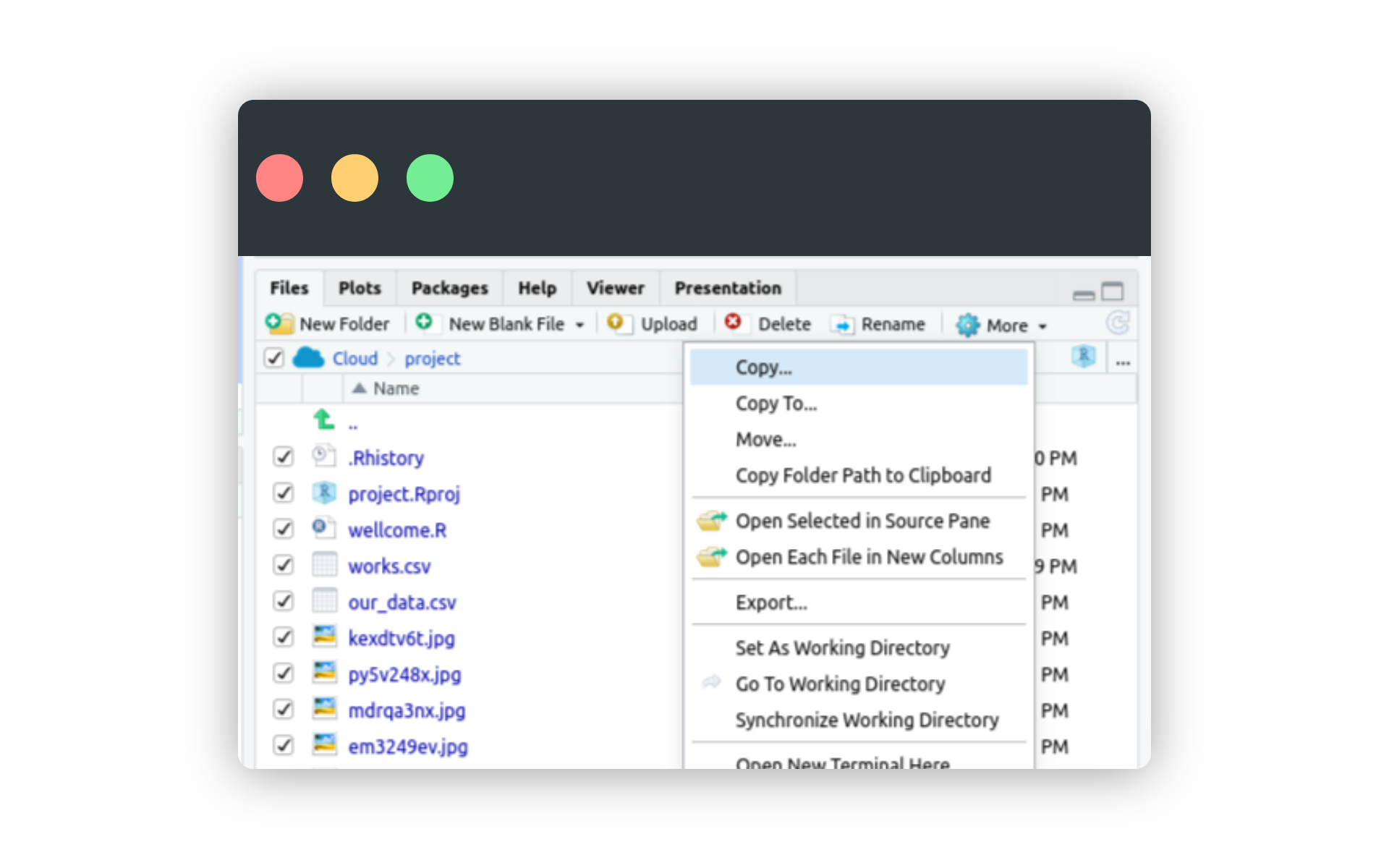
Po uruchomieniu funkcji getFilesFromWC(filtered_data) pliki powinny pobrać się do środowiska w Posit.cloud. Jeśli chcemy je pobrać na dysk, zaznaczamy pole checkbox przy ikonie Cloud i wybieramy opcję More > Export.
Pozyskane grafiki mają niewielkie rozmiary - jednak można temu zaradzić. Skany Wellcome Collection udostępniane są za pomocą protokołu IIIF (International Image Interoperability Framework) - ale o tym, jak z niego skorzystać, dowiemy się już na innej lekcji.
Podsumowanie
W tej lekcji pracowaliśmy z API Wellcome Collection. Udało nam się zautomatyzować pozyskiwanie danych o obiektach oraz ściąganie plików. Napisaliśmy dwie funkcje wykorzystujące pętle while i for oraz użyliśmy metod filtrowania danych dostępnych dzięki pakietowi dplyr.
Pracując z cyfrowymi zbiorami instytucji kultury i dziedzictwa nie jesteśmy skazani na korzystanie wyłącznie z gotowych narzędzi. Dlaczego nie mielibyśmy pisać własnych, lepiej dostosowanych do naszych celów? I dalej, nikt nie zabrania nam z nimi eksperymentować - nie zepsujemy API Wellcome Collection, jeśli nasze funkcje nie będą działać poprawnie.
Praca w Posit.cloud uwalnia nas od żmudnego przygotowywania środowiska R. Jeśli w trakcie pracy coś zepsujemy, nadpiszemy dane albo nasze funkcje po ostatniej edycji przestaną działać, możemy bardzo szybko zacząć od początku.
Wykorzystanie metod
R pozwala na budowanie własnych narzędzi, dostosowanych do konkretnych zadań czy to w pracy naukowej, czy eksploracji zbiorów muzeów, archiwów i bibliotek cyfrowych. Niekiedy wystarczy skorzystać z gotowych rozwiązań - przykładowo, w artykule Heritage Education and Research in Museums. Conceptual, Intellectual and Social Structure within a Knowledge Domain (2000–2019) autorzy korzystają z pakietów Bibliometrix i VOSviewer do przeprowadzenia analizy piśmiennictwa naukowego na temat badań i edukacji muzealnej. W innym przypadku własne narzędzia w R mogą pozwolić na bardziej efektywną pracę z katalogami, analizowanie danych o wizytach czy generowanie automatycznych raportów.
Pomysł na warsztat
Dwie lekcje poświęcone pracy z API Wellcome Collection to gotowa koncepcja warsztatu wprowadzającego do programowania w R. Można wykorzystać je jako część dłuższego kursu R lub w ramach pojedynczego warsztatu pokazującego możliwości, jakie daje wykorzystanie R w pracy ze zbiorami kultury i dziedzictwa.
W celu uzupełnienia tych lekcji o niezbędne wątki związane z pracą w R skorzystać można z podręcznika Jakuba Nowosada Elementarz programisty. Wstęp do programowania używając R, dostępnego na wolnej licencji CC BY.